自人类的祖先走出非洲后,杏耀平台人类经历的众多迁徙,基因混合以及自然选择等历史事件在基因组中留下了印记,形成了群体遗传结构。因此,推断群体遗传结构对于研究人群的演化历史至关重要,还能帮助校正全基因组关联分析(GWAS)中人群结构的混杂效应。2008年,Price等人发现了欧洲人群基因组中的一些区域会干扰人群群体遗传结构推断。但是,不同大洲的人群拥有不同的演化历史,并且近年来基因检测技术和人群基因组数据日新月异,亟需对这一问题进行系统性分析和更新。
近日,华中科技大学公共卫生学院王超龙教授团队基于千人基因组计划(1000 Genome Project)GRCh38版本人类参考基因组的高深度(30×)全基因组测序数据,通过系统分析,在欧洲,东亚,南亚,非洲人群中共找到27个干扰群体遗传结构推断的区域。在对人群进行主成分分析(PCA),或用ADMIXTURE软件推断群体遗传结构时,排除这27个基因组区域,
登录杏耀手机客户端,可得到更准确的推断结果。相关文章“Identification of genomic regions distorting population structure inference in diverse continental groups”(点击文末“阅读原文”下载PDF全文)发表在Quantitative Biology期刊上。
作者把对推断群体遗传结构具有异常贡献的基因组区域定义为“不寻常区域”。作者创建了一套生物信息分析流程用以确定这些“不寻常区域”,并分别应用于四个大洲的人群数据。
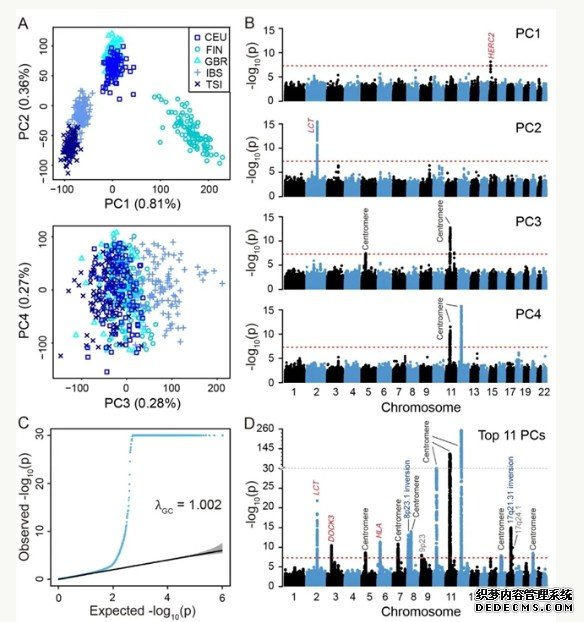
首先,对质控后的基因型数据进行PCA推断群体遗传结构,并得到每个变异位点对推断结果影响的大小,并据此确定对推断结果有异常影响的基因组区域。在去掉这些区域后,并基于剩余区域的基因型再次进行PCA。这个过程重复迭代多次,直到下一轮PCA不再出现新的异常区域。我们将每次迭代所找到的区域综合到一起,即为该大洲人群最终的“不寻常区域”(见图1)。作者在欧洲人群中发现了17个此类区域,东亚13个,南亚10个,非洲5个。随着大洲内部的遗传多样性增高,此类区域的数量减少。
作者对27个“不寻常区域”按其可能产生原因分为四类:区域位于染色体中心粒附近(13个),区域经历了染色体倒位(2个),区域受到自然选择(8个)和原因不明(4个)(见表1)。中心粒附近的区域由于对细胞的正常分裂有着至关重要的作用,其重组发生率很低,导致有着很长的连锁不平衡(LD)区域。另外染色体倒位和自然选择均可导致强LD。
接着,杏耀代理作者通过比较去除“不寻常区域”前后ADMIXTURE分析的结果发现,欧洲人群中的群体遗传结构推断受到的影响最大,去除后的结果更符合欧洲人的演化历史(见图2)。这也表明在遗传多样性较低的人群中,这些区域对群体遗传结构推断的结果产生的影响更为明显,与相关研究的结果一致。
Quantitative Biology (QB)期刊是由清华大学、北京大学、高教出版社联合创办的全英文学术期刊。QB主要刊登生物信息学、计算生物学、系统生物学、理论生物学和合成生物学的最新研究成果和前沿进展,并为生命科学与计算机、数学、物理等交叉研究领域打造一个学术水平高、可读性强、具有全球影响力的交叉学科期刊品牌。
QB期刊目前已被ESCI, Scopus, CSCD等国内外重要数据库收录。Citescore2021=4.6,预计明年将获得第一个影响因子(IF)。
由教育部主管、高等教育出版社主办的《前沿》(Frontiers)系列英文学术期刊,于2006年正式创刊,以网络版和印刷版向全球发行。系列期刊包括基础科学、生命科学、工程技术和人文社会科学四个主题,是我国覆盖学科最广泛的英文学术期刊群,其中13种被SCI收录,其他也被A&HCI、Ei、MEDLINE或相应学科国际权威检索系统收录,具有一定的国际学术影响力。系列期刊采用在线优先出版方式,保证文章以最快速度发表。





